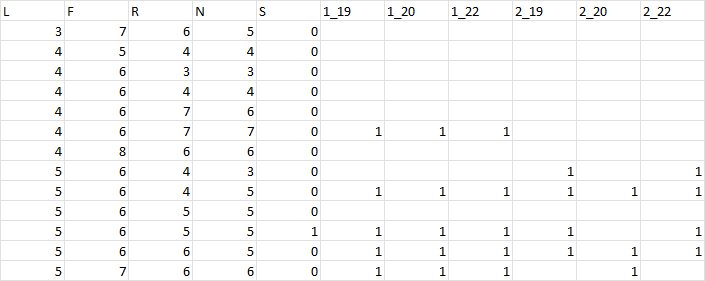
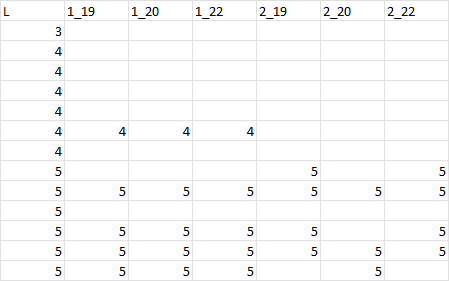
I have a data frame of presence (1) or absence (NA) of various plants found in different quadrats. I have 5 columns within the data frame of values that I would like to replace the 1's with. I'd like to end up with 5 different data frames.
Current Data Frame
This is a section of the data frame. Each row is a different plant. The columns L, F, R, N and S are the values I wish to replace the values win the last size columns ('1_19', '1_20' etc). Where there are blanks/NAs I want to keep them as blanks.
Expected Output
This is an example of the expected output for the L column. I would like to do this for the other columns too as separate dfs.
Data Frame
structure(list(Species = c("Conocephalum conicum", "Mnium hornum",
"Polytrichum formosum", "Oxalis acetosella", "Circaea lutetiana",
"Geum urbanum"), Common.Name = c("Great Scented Liverwort", "Swan's-neck Thyme-moss",
"Bank Haircap", "Wood Sorrel", "Enchanter's-nightshade", "Wood Avens"
), L = c(3L, 4L, 4L, 4L, 4L, 4L), F = c(7L, 5L, 6L, 6L, 6L, 6L
), R = c(6L, 4L, 3L, 4L, 7L, 7L), N = c(5L, 4L, 3L, 4L, 6L, 7L
), S = c(0L, 0L, 0L, 0L, 0L, 0L), Source = c("Hill et al., 2007",
"Hill et al., 2007", "Hill et al., 2007", "Hill et al., 1999",
"Hill et al., 1999", "Hill et al., 1999"), X1_19 = c(NA, NA,
NA, NA, NA, 1L), X1_20 = c(NA, NA, NA, NA, NA, 1L), X1_22 = c(NA,
NA, NA, NA, NA, 1L), X2_19 = c(NA_integer_, NA_integer_, NA_integer_,
NA_integer_, NA_integer_, NA_integer_), X2_20 = c(NA_integer_,
NA_integer_, NA_integer_, NA_integer_, NA_integer_, NA_integer_
), X2_22 = c(NA_integer_, NA_integer_, NA_integer_, NA_integer_,
NA_integer_, NA_integer_), X3_19 = c(NA, NA, NA, NA, NA, 1L),
X3_20 = c(NA_integer_, NA_integer_, NA_integer_, NA_integer_,
NA_integer_, NA_integer_), X3_22 = c(NA_integer_, NA_integer_,
NA_integer_, NA_integer_, NA_integer_, NA_integer_), X4_19 = c(NA_integer_,
NA_integer_, NA_integer_, NA_integer_, NA_integer_, NA_integer_
), X4_20 = c(NA_integer_, NA_integer_, NA_integer_, NA_integer_,
NA_integer_, NA_integer_), X4_22 = c(NA_integer_, NA_integer_,
NA_integer_, NA_integer_, NA_integer_, NA_integer_), X5_19 = c(NA_integer_,
NA_integer_, NA_integer_, NA_integer_, NA_integer_, NA_integer_
), X5_20 = c(NA_integer_, NA_integer_, NA_integer_, NA_integer_,
NA_integer_, NA_integer_), X5_22 = c(NA, NA, NA, NA, NA,
NA), X6_19 = c(NA, NA, NA, 1L, NA, NA), X6_20 = c(NA, NA,
NA, NA, 1L, NA), X6_22 = c(NA, NA, NA, 1L, NA, NA), X7_19 = c(NA_integer_,
NA_integer_, NA_integer_, NA_integer_, NA_integer_, NA_integer_
), X7_20 = c(NA, NA, NA, NA, 1L, NA), X7_22 = c(NA_integer_,
NA_integer_, NA_integer_, NA_integer_, NA_integer_, NA_integer_
), X8_19 = c(1L, NA, 1L, NA, NA, NA), X8_20 = c(NA_integer_,
NA_integer_, NA_integer_, NA_integer_, NA_integer_, NA_integer_
), X8_22 = c(1L, NA, 1L, NA, NA, NA), X9_19 = c(NA, 1L, NA,
NA, NA, NA), X9_20 = c(NA, 1L, NA, NA, NA, NA), X9_22 = c(NA,
1L, NA, NA, NA, NA)), row.names = c(NA, 6L), class = "data.frame")
CodePudding user response:
To get the desired output for all variables L..S, we could pivot_longer these variables, mutate across all variables starting with X, and use the grouping to split into 5 dataframes:
library(dplyr)
df |>
pivot_longer(L:S) |>
relocate(c(name, value), .before = "Species") |>
group_by(name) |>
mutate(across(starts_with("X"), ~ if_else(!is.na(.), value, NA_integer_))) |>
group_split()
Output:
[[1]]
# A tibble: 6 × 32
name value Species Common.Name Source X1_19 X1_20 X1_22 X2_19 X2_20 X2_22 X3_19 X3_20 X3_22 X4_19 X4_20 X4_22
<chr> <int> <chr> <chr> <chr> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int>
1 F 7 Conocephal… Great Scen… Hill … NA NA NA NA NA NA NA NA NA NA NA NA
2 F 5 Mnium horn… Swan's-nec… Hill … NA NA NA NA NA NA NA NA NA NA NA NA
3 F 6 Polytrichu… Bank Hairc… Hill … NA NA NA NA NA NA NA NA NA NA NA NA
4 F 6 Oxalis ace… Wood Sorrel Hill … NA NA NA NA NA NA NA NA NA NA NA NA
5 F 6 Circaea lu… Enchanter'… Hill … NA NA NA NA NA NA NA NA NA NA NA NA
6 F 6 Geum urban… Wood Avens Hill … 6 6 6 NA NA NA 6 NA NA NA NA NA
# … with 15 more variables: X5_19 <int>, X5_20 <int>, X5_22 <int>, X6_19 <int>, X6_20 <int>, X6_22 <int>,
# X7_19 <int>, X7_20 <int>, X7_22 <int>, X8_19 <int>, X8_20 <int>, X8_22 <int>, X9_19 <int>, X9_20 <int>,
# X9_22 <int>
[[2]]
# A tibble: 6 × 32
name value Species Common.Name Source X1_19 X1_20 X1_22 X2_19 X2_20 X2_22 X3_19 X3_20 X3_22 X4_19 X4_20 X4_22
<chr> <int> <chr> <chr> <chr> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int>
1 L 3 Conocephal… Great Scen… Hill … NA NA NA NA NA NA NA NA NA NA NA NA
2 L 4 Mnium horn… Swan's-nec… Hill … NA NA NA NA NA NA NA NA NA NA NA NA
3 L 4 Polytrichu… Bank Hairc… Hill … NA NA NA NA NA NA NA NA NA NA NA NA
4 L 4 Oxalis ace… Wood Sorrel Hill … NA NA NA NA NA NA NA NA NA NA NA NA
5 L 4 Circaea lu… Enchanter'… Hill … NA NA NA NA NA NA NA NA NA NA NA NA
6 L 4 Geum urban… Wood Avens Hill … 4 4 4 NA NA NA 4 NA NA NA NA NA
# … with 15 more variables: X5_19 <int>, X5_20 <int>, X5_22 <int>, X6_19 <int>, X6_20 <int>, X6_22 <int>,
# X7_19 <int>, X7_20 <int>, X7_22 <int>, X8_19 <int>, X8_20 <int>, X8_22 <int>, X9_19 <int>, X9_20 <int>,
# X9_22 <int>
[[3]]
# A tibble: 6 × 32
name value Species Common.Name Source X1_19 X1_20 X1_22 X2_19 X2_20 X2_22 X3_19 X3_20 X3_22 X4_19 X4_20 X4_22
<chr> <int> <chr> <chr> <chr> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int>
1 N 5 Conocephal… Great Scen… Hill … NA NA NA NA NA NA NA NA NA NA NA NA
2 N 4 Mnium horn… Swan's-nec… Hill … NA NA NA NA NA NA NA NA NA NA NA NA
3 N 3 Polytrichu… Bank Hairc… Hill … NA NA NA NA NA NA NA NA NA NA NA NA
4 N 4 Oxalis ace… Wood Sorrel Hill … NA NA NA NA NA NA NA NA NA NA NA NA
5 N 6 Circaea lu… Enchanter'… Hill … NA NA NA NA NA NA NA NA NA NA NA NA
6 N 7 Geum urban… Wood Avens Hill … 7 7 7 NA NA NA 7 NA NA NA NA NA
# … with 15 more variables: X5_19 <int>, X5_20 <int>, X5_22 <int>, X6_19 <int>, X6_20 <int>, X6_22 <int>,
# X7_19 <int>, X7_20 <int>, X7_22 <int>, X8_19 <int>, X8_20 <int>, X8_22 <int>, X9_19 <int>, X9_20 <int>,
# X9_22 <int>
[[4]]
# A tibble: 6 × 32
name value Species Common.Name Source X1_19 X1_20 X1_22 X2_19 X2_20 X2_22 X3_19 X3_20 X3_22 X4_19 X4_20 X4_22
<chr> <int> <chr> <chr> <chr> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int>
1 R 6 Conocephal… Great Scen… Hill … NA NA NA NA NA NA NA NA NA NA NA NA
2 R 4 Mnium horn… Swan's-nec… Hill … NA NA NA NA NA NA NA NA NA NA NA NA
3 R 3 Polytrichu… Bank Hairc… Hill … NA NA NA NA NA NA NA NA NA NA NA NA
4 R 4 Oxalis ace… Wood Sorrel Hill … NA NA NA NA NA NA NA NA NA NA NA NA
5 R 7 Circaea lu… Enchanter'… Hill … NA NA NA NA NA NA NA NA NA NA NA NA
6 R 7 Geum urban… Wood Avens Hill … 7 7 7 NA NA NA 7 NA NA NA NA NA
# … with 15 more variables: X5_19 <int>, X5_20 <int>, X5_22 <int>, X6_19 <int>, X6_20 <int>, X6_22 <int>,
# X7_19 <int>, X7_20 <int>, X7_22 <int>, X8_19 <int>, X8_20 <int>, X8_22 <int>, X9_19 <int>, X9_20 <int>,
# X9_22 <int>
[[5]]
# A tibble: 6 × 32
name value Species Common.Name Source X1_19 X1_20 X1_22 X2_19 X2_20 X2_22 X3_19 X3_20 X3_22 X4_19 X4_20 X4_22
<chr> <int> <chr> <chr> <chr> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int>
1 S 0 Conocephal… Great Scen… Hill … NA NA NA NA NA NA NA NA NA NA NA NA
2 S 0 Mnium horn… Swan's-nec… Hill … NA NA NA NA NA NA NA NA NA NA NA NA
3 S 0 Polytrichu… Bank Hairc… Hill … NA NA NA NA NA NA NA NA NA NA NA NA
4 S 0 Oxalis ace… Wood Sorrel Hill … NA NA NA NA NA NA NA NA NA NA NA NA
5 S 0 Circaea lu… Enchanter'… Hill … NA NA NA NA NA NA NA NA NA NA NA NA
6 S 0 Geum urban… Wood Avens Hill … 0 0 0 NA NA NA 0 NA NA NA NA NA
# … with 15 more variables: X5_19 <int>, X5_20 <int>, X5_22 <int>, X6_19 <int>, X6_20 <int>, X6_22 <int>,
# X7_19 <int>, X7_20 <int>, X7_22 <int>, X8_19 <int>, X8_20 <int>, X8_22 <int>, X9_19 <int>, X9_20 <int>,
# X9_22 <int>
CodePudding user response:
Using dplyr, this should work :
L=df%>%
rowwise()%>%
mutate_at(.vars=colnames(df)[9:35],funs(replace(., which(.==1), L)))
CodePudding user response:
And here's a relatively simple version using purrr to get your 5 data frames (as list elements). The selection of the columns X1_... can of course be changed to your liking.
check_names <- c("L", "F", "R", "N", "S")
library(tidyverse)
check_names |>
map(\(current_name) df |>
select(-setdiff(check_names, current_name)) |>
rowwise() |>
mutate(across(matches("^X\\d _\\d"), ~ifelse(is.na(.), ., .data[[current_name]]))) |>
ungroup()) |>
set_names(check_names)
$L
# A tibble: 6 × 31
Species Common.Name L Source X1_19 X1_20 X1_22 X2_19 X2_20 X2_22 X3_19 X3_20 X3_22 X4_19 X4_20 X4_22 X5_19 X5_20 X5_22 X6_19 X6_20 X6_22 X7_19 X7_20 X7_22 X8_19 X8_20 X8_22 X9_19 X9_20 X9_22
<chr> <chr> <int> <chr> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <lgl> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int>
1 Conocephalum conicum Great Scented Liverwort 3 Hill et al., 2007 NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA 3 NA 3 NA NA NA
2 Mnium hornum Swan's-neck Thyme-moss 4 Hill et al., 2007 NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA 4 4 4
3 Polytrichum formosum Bank Haircap 4 Hill et al., 2007 NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA 4 NA 4 NA NA NA
4 Oxalis acetosella Wood Sorrel 4 Hill et al., 1999 NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA 4 NA 4 NA NA NA NA NA NA NA NA NA
5 Circaea lutetiana Enchanter's-nightshade 4 Hill et al., 1999 NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA 4 NA NA 4 NA NA NA NA NA NA NA
6 Geum urbanum Wood Avens 4 Hill et al., 1999 4 4 4 NA NA NA 4 NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA
$F
# A tibble: 6 × 31
Species Common.Name F Source X1_19 X1_20 X1_22 X2_19 X2_20 X2_22 X3_19 X3_20 X3_22 X4_19 X4_20 X4_22 X5_19 X5_20 X5_22 X6_19 X6_20 X6_22 X7_19 X7_20 X7_22 X8_19 X8_20 X8_22 X9_19 X9_20 X9_22
<chr> <chr> <int> <chr> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <lgl> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int>
1 Conocephalum conicum Great Scented Liverwort 7 Hill et al., 2007 NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA 7 NA 7 NA NA NA
2 Mnium hornum Swan's-neck Thyme-moss 5 Hill et al., 2007 NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA 5 5 5
3 Polytrichum formosum Bank Haircap 6 Hill et al., 2007 NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA 6 NA 6 NA NA NA
4 Oxalis acetosella Wood Sorrel 6 Hill et al., 1999 NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA 6 NA 6 NA NA NA NA NA NA NA NA NA
5 Circaea lutetiana Enchanter's-nightshade 6 Hill et al., 1999 NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA 6 NA NA 6 NA NA NA NA NA NA NA
6 Geum urbanum Wood Avens 6 Hill et al., 1999 6 6 6 NA NA NA 6 NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA
$R
# A tibble: 6 × 31
Species Common.Name R Source X1_19 X1_20 X1_22 X2_19 X2_20 X2_22 X3_19 X3_20 X3_22 X4_19 X4_20 X4_22 X5_19 X5_20 X5_22 X6_19 X6_20 X6_22 X7_19 X7_20 X7_22 X8_19 X8_20 X8_22 X9_19 X9_20 X9_22
<chr> <chr> <int> <chr> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <lgl> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int>
1 Conocephalum conicum Great Scented Liverwort 6 Hill et al., 2007 NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA 6 NA 6 NA NA NA
2 Mnium hornum Swan's-neck Thyme-moss 4 Hill et al., 2007 NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA 4 4 4
3 Polytrichum formosum Bank Haircap 3 Hill et al., 2007 NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA 3 NA 3 NA NA NA
4 Oxalis acetosella Wood Sorrel 4 Hill et al., 1999 NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA 4 NA 4 NA NA NA NA NA NA NA NA NA
5 Circaea lutetiana Enchanter's-nightshade 7 Hill et al., 1999 NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA 7 NA NA 7 NA NA NA NA NA NA NA
6 Geum urbanum Wood Avens 7 Hill et al., 1999 7 7 7 NA NA NA 7 NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA
$N
# A tibble: 6 × 31
Species Common.Name N Source X1_19 X1_20 X1_22 X2_19 X2_20 X2_22 X3_19 X3_20 X3_22 X4_19 X4_20 X4_22 X5_19 X5_20 X5_22 X6_19 X6_20 X6_22 X7_19 X7_20 X7_22 X8_19 X8_20 X8_22 X9_19 X9_20 X9_22
<chr> <chr> <int> <chr> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <lgl> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int>
1 Conocephalum conicum Great Scented Liverwort 5 Hill et al., 2007 NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA 5 NA 5 NA NA NA
2 Mnium hornum Swan's-neck Thyme-moss 4 Hill et al., 2007 NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA 4 4 4
3 Polytrichum formosum Bank Haircap 3 Hill et al., 2007 NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA 3 NA 3 NA NA NA
4 Oxalis acetosella Wood Sorrel 4 Hill et al., 1999 NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA 4 NA 4 NA NA NA NA NA NA NA NA NA
5 Circaea lutetiana Enchanter's-nightshade 6 Hill et al., 1999 NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA 6 NA NA 6 NA NA NA NA NA NA NA
6 Geum urbanum Wood Avens 7 Hill et al., 1999 7 7 7 NA NA NA 7 NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA
$S
# A tibble: 6 × 31
Species Common.Name S Source X1_19 X1_20 X1_22 X2_19 X2_20 X2_22 X3_19 X3_20 X3_22 X4_19 X4_20 X4_22 X5_19 X5_20 X5_22 X6_19 X6_20 X6_22 X7_19 X7_20 X7_22 X8_19 X8_20 X8_22 X9_19 X9_20 X9_22
<chr> <chr> <int> <chr> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <lgl> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int> <int>
1 Conocephalum conicum Great Scented Liverwort 0 Hill et al., 2007 NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA 0 NA 0 NA NA NA
2 Mnium hornum Swan's-neck Thyme-moss 0 Hill et al., 2007 NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA 0 0 0
3 Polytrichum formosum Bank Haircap 0 Hill et al., 2007 NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA 0 NA 0 NA NA NA
4 Oxalis acetosella Wood Sorrel 0 Hill et al., 1999 NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA 0 NA 0 NA NA NA NA NA NA NA NA NA
5 Circaea lutetiana Enchanter's-nightshade 0 Hill et al., 1999 NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA 0 NA NA 0 NA NA NA NA NA NA NA
6 Geum urbanum Wood Avens 0 Hill et al., 1999 0 0 0 NA NA NA 0 NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA